Auch die Seqeunzierung von Corona-Viren erfolgt mit dieser Methode.
Next Generation Sequencing (NGS)
Erfolgreiche Methode zur Aufklärung von lebensmittelbedingten Krankheitsausbrüchen
Die Erbinformation eines Bakterienstammes – der genetische Code – ist so individuell wie der menschliche Fingerabdruck. Durch Entschlüsseln dieses Codes wird ein Bakterienstamm eindeutig identifiziert. Das „Next Generation Sequencing“ (NGS, deutsch: Sequenzierung der nächsten Generation) ist eine schnelle Methode, die gesamte Erbinformation beispielsweise eines Krankheitserregers zu entschlüsseln. Ist der genetische Code eines Krankheitserregers aus Patienten bekannt und vergleicht man diesen mit Bakterienstämmen aus verdächtigen Lebensmitteln, können daraus Informationen auf eine mögliche Verbindung gewonnen werden. Durch weitere epidemiologische Untersuchungen kann die NGS, als ein Teil der Aufklärung von lebensmittelbedingten Krankheitsausbrüchen, einen wertvollen Beitrag liefern und weitere Erkrankungen verhindern.
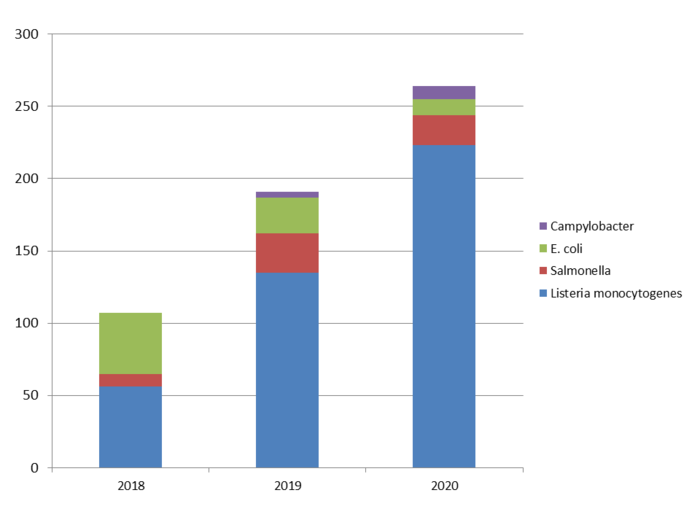
Seit Januar 2018 wird NGS am Standort Braunschweig des Lebensmittel- und Veterinärinstituts (LVI) Braunschweig/Hannover zur Sequenzierung von Bakterienisolaten aus Lebensmitteln und der Produktionsumgebung von Lebensmittelunternehmen eingesetzt. Seither wurden rund 570 Isolate (Stand Dezember 2020), vor allem von Listeria monocytogenes, untersucht.
| Ein Beispiel
Nach Verzehr eines Brötchens mit Räucherlachs erkrankte ein Verbraucher an Listeriose. Eine Probe des Räucherlachses wurde vom zuständigen kommunalen Veterinäramt an das LAVES-Institut für Fische und Fischereierzeugnisse gesandt. Dort wurde ein sehr hoher Gehalt an Listeria monocytogenes nachgewiesen. Am LVI Braunschweig erfolgte anschließend die Sequenzierung des Genoms. Das Niedersächsische Landesgesundheitsamt untersuchte parallel eine Probe des Erkrankten ebenfalls mittels NGS. Sowohl das Lebensmittelisolat als auch das Isolat des Erkrankten konnten durch die zuständigen Bundeseinrichtungen (RKI und BfR) einem bereits bekannten Krankheitsgeschehen zugeordnet werden. Damit wurden die Listeriosefälle in diesem Cluster erstmals einem bestimmten Lebensmittel zugeordnet. |
Ziel ist es, das NGS auch im Bereich der pathogenen (krankmachenden) Viren zu etablieren. Das Spektrum soll sowohl lebensmittelassoziierte Viren (zum Beispiel Noroviren, Hepatits-A-Viren) als auch Viren aus dem veterinärmedizinischen Bereich (zum Beispiel Geflügelgrippe) umfassen. Auch die Sequenzierung des Sars-CoV-2-Virus ist mit dieser Methode möglich.
Bei der NGS-Untersuchung im Bereich der Viren handelt es sich selten um die Untersuchung eines Einzel-Isolats mittels Gesamtgenom-Sequenzierung. Vielmehr kommt hier eine Metagenom-Analyse zum Einsatz. Vorteil ist, dass nicht nur nach einem bestimmten Virus gesucht wird, sondern auch bisher nicht bekannte Viren (zum Beispiel Schmallenberg-Virus) identifiziert werden können. Auch Mikroorganismen, die nur schwer oder gar nicht zu kultivieren sind, lassen sich mit der Metagenom-Analyse nachweisen.
Weitere Informationen
Bundesinstitut für Risikobewertung: Erbmaterial von Erregern vergleichen, um Krankheitsausbrüche aufzuklären(PDF)




 English
English